|
:: 게시판
:: 이전 게시판
|
- 자유 주제로 사용할 수 있는 게시판입니다.
- 토론 게시판의 용도를 겸합니다.
통합규정 1.3 이용안내 인용"Pgr은 '명문화된 삭제규정'이 반드시 필요하지 않은 분을 환영합니다.법 없이도 사는 사람, 남에게 상처를 주지 않으면서 같이 이야기 나눌 수 있는 분이면 좋겠습니다."
20/11/17 01:38
재미있네요~ 예전에 잠깐 bioinformatics에 관심을 가졌는데 그 때만 해도 java library밖에 없었는데 이젠 R을 사용하는군요
20/11/17 01:51
single cell이 최근 핫하긴 하죠. DNA는 암세포나 특이하게 변이에서 오는 병이 아닌 이상 한 사람이 평생 같은 정보를 가지고 사는데에 비해서 RNA는 세포마다 그리고 현 몸 상황에 따라서 계속 바뀌기 때문에 훨씬 더 많이 연구될거라 봅니다.
이상적인 상황은 모두가 DNA정보를 가지고 있고 그걸 바탕으로 병이나 문제가 있으면 RNA를 이용해 원인을 진단하고 치료방향을 찾아보는 쪽으로 가겠죠.
20/11/17 14:31
DNA에서 현재 필요한 RNA만 만들어 내게 하는 기능을 RNA polymerase가 담당하나요? transcription을 제어하는 인자는 무엇인가요?
20/11/17 14:57
(수정됨) 어느 단계에서 어떻게 제어하느냐에 따라 방법이 여러가지라서 자세히 설명하기엔 아마 따로 글이 필요할거 같고 일단 짧게나마 설명을 해보자면
- DNA가 크로마틴으로 패킹이 되어있으면 아예 접근이 불가능합니다 - DNA가 접근이 가능해도 methlyation으로 막혀있으면 읽을수 없습니다. 이것만을 따로 연구하는 methyl-seq이 있습니다 - DNA를 읽을수 있으면 이제 유전인자 마다 방식이 다릅니다. 유전인자의 앞부분의 염기서열은 프로모터/사일렌서/언핸서 단백질들이 붙을수 있고 그 뒷부분이 실제로 transcription이 되는 염기서열입니다. 그래서 저 앞부분에 어떤 단백질이 붙을수 있느냐에 따라 유전인자의 활동이 올라가거나 내려가거나 아예 멈추거나 합니다. - 같은 유전인자 안에서도 스플라이싱이 어떻게 되느냐에 따라서 또 다른 단백질이 만들어질수 있습니다. 혹시 관심이 있으시다면 저런걸 공부하는걸 통틀어서 epigenetics라고 합니다. 실제론 저 유전인자 하나와 다른 단백질 하나의 메커니즘을 설명하는데 눈문 하나씩 나오니까 훨씬 복잡합니다만은 대부분 저런식으로 제어가 됩니다.
20/11/17 02:06
저도 기본적으로 생물학 화학 문맹인데, 제가 이 분야에서 강렬하게 매력을 느낀 부분은 '일단 행렬로 바꾼 후에는 수학적 기법으로 모든 것을 해석한다' 는 점이었어요. 저기서부터는 관련 연구자들도 대부분 수학자나 통계학 전공자들이더라고요.
20/11/17 06:52
이과에서 가장 이과적이지 않은 분야가 생물학이라고 생각합니다. 이런 저런 현상을 딱 부러지게 설명하기가 좀 그런게 많이 있거든요. 하다못해 실험을 할때 재료를 준비할때도 꼭 여유분을 준비해야지, 안그럼 실험 나중에 모자라서 멘붕오죠. 건축학을 공부한 우리 와이프는 그 얘기를 듣더니 꼭 토목공사라고 쓰고 (노가다)랑 비슷하다고 하더군요.
바이오피직스/스탯/인포메틱스 이런 분야들은 보통 바이오XXX 에서 XXX 분야의 전공자들이 생물학이라 말하는 분야들을 공부한 분들이 하시죠. 왜냠 생물학 하는 분들이 그 XXX 를 공부하기엔 너무도 많은 시간이 걸리기 때무에 ROI 가 나오지 않아서죠 (2000년 중반까지 이야기입니다. 요즘 생물학 하는 스마트한 분들 많아서 다를겁니다).
20/11/17 23:08
그게 참 딜레마인 것 같더라고요. 전공 지식을 많이 쌓자니 수학이 약해서 남들이 만든 툴을 수동적으로 받아 쓰기만 해야하고, 수학을 파자니 해석을 못하고. 뭐 우리가 몰라서 그렇지 그런 분야가 생물학 말고도 또 있긴 하겠죠.
20/11/17 02:22
요즘 겉만 생물학자이지 컴싸에 더 가까운 computational biology나 biostatitics가 뜨고있죠.
마지막에 보이는 UMAP이 제일 최근에 나온 dimensionality reduction 알고리즘인데 그전에 쓰이던 pca 나 t-sne보다 더 좋다고 합니다. 보통 다차원 데이터를 2차원 시각화할때 쓰입니다.
20/11/17 04:38
그래서 저는 오히려 걱정이긴 합니다.
다 Biostat이나 Comp bio쪽으로 하려니 정작 실험하는 사람들이 많이 없어서요. 그래서 CS쪽은 배우기만 하고 실험쪽에 남아야 하나 생각도 듭니다
20/11/17 04:48
실험은 사실 FACS 정도만 할줄 알면 되는데 문제는 실험에 익숙하지 않으면 그 많은 데이터를 봐도 뭐가 중요한지 알기가 어렵다는 것이죠. 온톨로지 수준에서 중요한 것도 있지만 온톨로지 분석은 유전자 숫자 싸움이라는 한계도 있죠. 실험 학문과 실험 논문에 익숙하지 않으면, 수는 적지만 분명히 중요한 생물학적 의미를 가지는 신호전달경로를 놓칠 수 있다고 봅니다.
20/11/17 04:42
(수정됨) scRNA-seq을 활용한 논문을 이번달에 투고하려고 한데, 이런 글을 보니 반갑네요.
저는 생물정보학은 잘 몰라서 DGIST의 김종경 교수님의 도움을 많이 받았습니다. 서울대 황대희 교수님과 이번에 카이스트에 부임한 박종은 교수님도 scRNA-seq 잘하시는 것으로 유명하죠. 기회가 되면 저 기술을 이용해서 어떤 연구들이 가능한지 소개하는 글을 써보겠습니다 흐흐
20/11/17 14:12
꼭 써주십시요.
써주시면 제가 방학기간동안 Next Generation Sequencing 과 Kit development에 대해서 써 보겠습니다
20/11/17 06:10
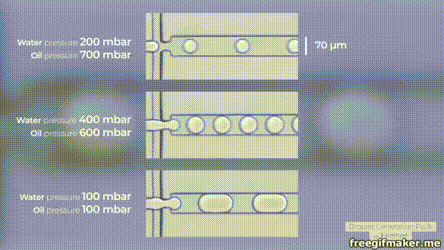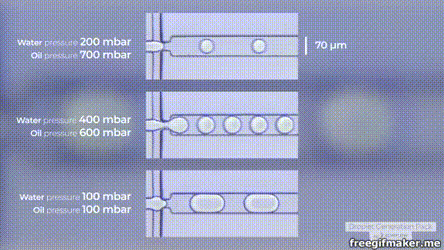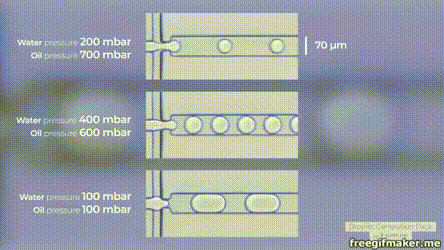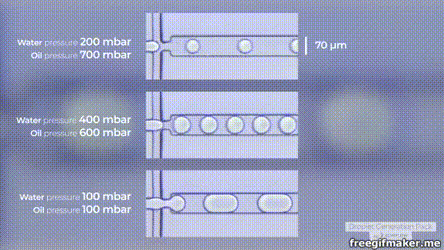
오래전 폭닥할때 제가 있던 랩에서 저 Emulsion droplet PCR 초기컨셉으로 열심히 했었습니다. Oild droplet 안에 RNA/Enzyme/dNTP 등등 섞어서 저걸로 Artificial Cell을 만들려고 했었죠. 전에 썼지만 우리 랩 출신들은 보스가 저걸로 노벨상 또 한번 받지않을까 기대했습니다. (왜 과거형이냐면 요즘 제가 연구를 그만둔지 괘 되어서 그후 일들은 잘 모릅니다). 제가 석사할때 90년대 초, RNA Splicling 이란 분야가 핫했었고(주로 Antibody의 생성에 관하여), 그후 휴먼 지놈 프로젝때문에 RNA Editing 이란 분야와 Transcriptome/Orfeom 이란 분야가 emerging 하였죠.
PCA 분석은 MS 로 분석하는게 아닌 젤 이미지로 분석하는 Proteomics Analysis Package (2-D DIGE from Amerhsame/GE Healthcare)에서 쓰여졌습니다. 한장의 젤에 수백개의 서로 다른 시그날을 가진 프로테인 스팟들을 그룹핑하고 현격한 차이가 있는 후보들을 추려내는데 쓰였죠. 제가 연구 그만하고 GE Healthcare로 옮겨서 했던 일이 FAS 였는데, 마침 저 소프트웨어 트래이닝 담당이라서 생각이 나네요. 오랜만에 옛생각이 나는 글 올려주셔서 감사합니다.
20/11/17 10:54
저도 신경발생 전공했고 박사과정하면서 나름 발생학을 깊이 안다고 생각했는데 보여주신 기술과 자료분석은 정말 another level이네요. 좋은 공부되었습니다. 감사합니다.
20/11/17 12:02
생물 쪽은 figure가 이쁘네요. 재료나 고체물리쪽에서 원자구조는 vesta 같은거로 많이 그리는데, 저런 그림 그려주는 전용 프로그램이 있는거겠죠? 다 일러스트레이터로 작업하지는 않았을 것 같은데...맨 밑에 figure a, b, c, d 저 데이터는 뭘로 피팅한걸까요? 오리진은 아닌거 같은데...
20/11/17 12:27
R의 Seurat 패키지로 그린 것 같습니다 (https://www.nature.com/articles/s41587-020-0469-4).
사실 저는 그릴 줄 모르는데, 같이 일하는 후배가 그리는 걸 자주 봤어요. 저도 올해가 가기 전에 배워볼라고 합니다 흐흐
20/11/17 14:26
모든 RNA가 기능이 다 밝혀진 것은 아닐텐데. ontology는 어떻게 정리된 것인지 궁금합니다. 대충 알려진 것들만 모아놓은 것 아닌가요?
기능이 없지 않지만 ontology에 등록되어 있지 않은 경우 새로운 RNA의 기능을 증명? 해서 ontology에 넣고 싶다면 어떻게 해야하나요?
20/11/17 15:16
그건 제가 전공자가 아니라서 다른 분께서 대답해주시길 원합니다만, 새로이 ontology 에 추가하는 것또한 주요 연구 주제인 것 같습니다.
20/11/17 14:56
중간에 수학적인 모델은 문송해서 잘 이해가 안갑니다만, 그러니까 세포를 하나씩 뜯어볼 수 있고, 거기에 수학적 모델을 적용시키면 유전정보에 대한 분포도를 얻어낼 수 있다는 말씀이시죠? 흥미롭네요. 제가 좋아하는 컴퓨터 게임의 '오픈소스' 같은 개념으로 공유가 그렇게 연구기관 사이에서 쉽게쉽게 될 수 있다면, 당연히 '오픈소스 모딩'도 등장할 수 있을것 같군요 와아... 신기해라!
20/11/17 15:18
문과시면서 이렇게 쉽게 개요를 이해하시니 대단하십니다. 이 분야는 상당 수준까지는 오픈소스 개념으로 공유가 되는 것 같습니다. 그래서 더욱 신기해요!
20/11/17 17:00
석사 초반에 Bioinfomatics할 때
yeast가지고 in situ hybridization을 해서 Nouthern Blotting으로 RNA sequence 필름으로 뽑아서 일일이 하나씩 매칭해서 적은 다음 BLASTing해서 매칭 Sequence를 PC로 돌릴때 생각하면 정말 상전벽해입니다. 사실 지금 쓰신 기술의 경우 Cell 3D bioprinting 할때도 씌입니다.
|
||||||||||